Alle Proteine, die Zellen für ihre jeweiligen Aufgaben nutzen, haben einen gemeinsamen Ursprung: das Genom. Im Genom sind, in Form von DNA-Sequenzen, «Bauanleitungen», genannt Gene, für all diese Proteine gespeichert. Das zentrale Dogma der Molekularbiologie gibt Auskunft darüber, welche Prozesse für die Herstellung von Proteinen benötigt wird. Die Proteinsynthese wird durch zwei eng miteinander verknüpften biochemischen Prozessen ausgeführt. Im ersten Teilprozess erfolgt die Umschreibung der Erbinformation der DNA mit Hilfe einer RNA-Polymerase zu einer Boten-RNA (engl.: messenger-RNA, mRNA). Dieser Prozess wird Transkription genannt. Der zweite Teilrozess beinhaltet die Übertragung der mRNA in eine Aminosäurenfolge an den Ribosomen. Die Aminosäuren werden mithilfe der Transport-RNA (engli.: transfer RNA, tRNA) zum Ribosom transportiert. Dieser Prozess wird Translation genannt.
3.1 – Aufbau von DNA:
DNA (Desoxyribonucleinsäure) ist ein lineares, aus zwei, zu einer Doppelhelix gewundenen, Strängen bestehendes Makromolekül. Jeder Strang besteht aus zwei Einheiten, die immer abwechselnd aneinandergeknüpft sind: Phosphat-Einheiten und Zucker-Basen-Einheiten (Abb. 3.1). Die Zucker-Einheit besteht aus Desoxyribose. An jeder Desoxyribose-Einheit hängt eine Basen-Einheit, nämlich eine von vier verschiedenen Nucleobasen. Die vier natürlichen Nucleobasen sind: Adenin, Thymin, Guanin und Cytosin, oder kurz A, T, G und C. Information wird in der DNA durch die Abfolge dieser Nucleobasen gespeichert. Für die Proteinsynthese bilden jeweils drei aufeinanderfolgende Nucleobasen eine Informationseinheit, ein sogenanntes Codon. Jedes Codon codiert während der Translation für eine ganz bestimmte Aminosäure. Die Abfolge auf dem DNA-Strang bestimmt also den Aufbau des Proteins.
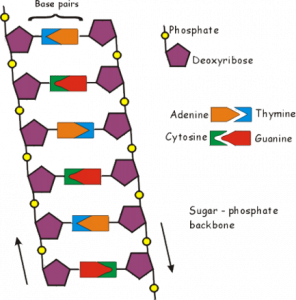
Abbildung 3.1 – Struktur des DNA-Moleküls. Das Desoxyribose-Phosphat hat Basenseitenketten, welche über Wasserstoffbrücken mit der Base des anderen Strangs verbunden sind. Adenin macht immer eine Basenpaarung mit Thymin, Cytosin mit Guanin. (Illustration von Beverly High School, 2005, Psionica)
3.2 – Aufbau von RNA:
RNA (Ribonucleinsäure) ist ein Molekül, welches sehr ähnlich zur DNA ist. Es gibt allerdings einige wesentliche Unterschiede:
- RNA besteht aus nur einem Strang.
- Anstatt der Nucleobase Thymin existiert in der RNA die Nucleobase Uracil, welche dem Thymin aber sehr ähnlich ist.
- Anstatt Desoxyribose ist in der RNA Ribose als Zucker verbaut.
Die Ribose im Zucker-Phosphat-Rückgrat führt dazu, dass die vorhandene OH-Gruppe mit der Phosphatgruppe des Rückgrates reagieren und somit ein Strangbruch induzieren kann. Dies führt dazu, dass die Stabilität von RNA geringer ist als die der DNA. Sie eignet sich aber hervorragend als kurzfristige Kopie von Genen, da sie schnell und unkompliziert wieder abgebaut werden kann. Die Halbwertszeit von RNA liegt normalerweise im Bereich von Stunden, wobei die DNA eine Halbwertszeit von Hunderten von Jahren hat. RNA, welche die kopierte Information aus Genen enthält, wird messengerRNA, kurz mRNA, genannt und spielt eine wesentliche Rolle in der Transkription und Translation.
3.3 – Struktur von Genen:
Wir betrachten in diesem Buch die Struktur von Genen. Generell muss berücksichtigt werden, ob prokaryotische oder eukaryotische Gene betrachtet werden, denn es gibt beim Aufbau einige Unterschiede. Wir betrachten in diesem Buch meistens prokaryotische Gene. Die Unterschiede zu den Eukaryoten können aber meist ohne grossen Aufwand übertragen werden. Sei das ein unterschiedlicher Aufbau der RNA-Polymerase oder dass es Transkriptionsfaktoren gibt, die an die RNA-Polymerase binden. Wir werden das Modell auf prokaryotischen Transkription aufbauen. Damit die Transkriptions-Maschinerie Gene auch erkennen kann und diese auch korrekt abliest, haben alle Gene eine gemeinsame Struktur (Abb. 3.2).
Jedes Gen enthält einen codierenden Teil, der in den meisten Fällen für den Bauplan des Proteins codiert. Dieser Teil des Gens wird während der Transkription „abgelesen“ und trägt daher den Namen Open-Reading-Frame, oder kurz ORF. Vor dem ORF befindet sich eine Gensequenz, die den Beginn des Gens anzeigt, die sogenannte Promotor-Region. Diese Region ist essentiell für den modellierten Vorgang. Außerdem kann sich zwischen dem Promotor und dem ORF noch eine Operator-Region befinden, welche die Transkription zusätzlich beeinflussen kann. Am Ende jedes Gens befindet sich eine Terminator-Region, welche das Ende eines Gens anzeigt.
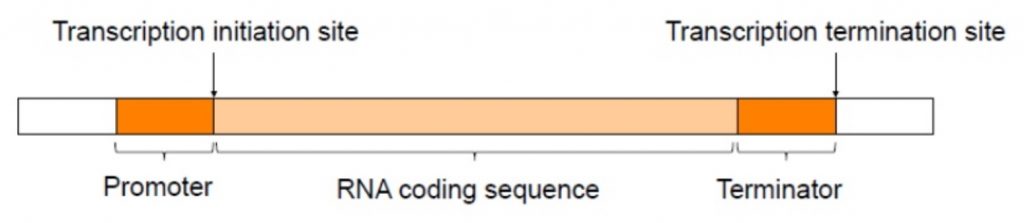
Abbildung 3.2 – Aufbau eines Gens bestehend aus dem Promotor, an welchem die RNA-Polymerase bindet, die RNA-codierende Sequenz, die während der Translation in Aminosäuren umgewandelt wird und der Terminator, der das Ende der Transkription markiert. (Illustration von Molecular Biology, Kayeen Vadakkan, Department of Biotechnology, St. Mary’s College, Thrissur)
3.4 – Promotoren
Die Promotor-Region markiert den Anfang eines Gens, indem sie die Bindungstelle für die RNA-Polymerase ist, dem Enzym, das während der Transkription den Open-Reading-Frame abliest.
Da es in prokraryotischen Zellen eine sehr hohe Anzahl an Genen gibt, aber nur eine bestimmte Art zu mRNA-Polymerase, müssen alle Promotoren ähnlich aufgebaut sein, damit sie alle von demselben Molekül erkannt werden können. Die bakterielle RNA-Polymerase bindet verschiedene Sigmafaktoren, die dann bestimmte Konsensus-Sequenzen erkennen und dadurch verschiedene Gensets ablesen. Ein Beispiel für eine Konsensussequenz ist die Pribnow-Box, eine Adenin- und Thymin-reiche Region, die in bakteriellen Promotoren vorkommt und vom Sigmafaktor 70 erkannt wird, welcher der wichtigste Sigmafaktor unter normalen Wachstumsbedingungen bei Prokaryoten ist.
Promotoren dienen aber nicht nur als einfacher „Startpunkt“ eines Gens. Sie haben eine weitere Funktion: Sie können helfen die Genexpression zu regulieren. Hierbei lassen sich 2 Promotor-Typen unterscheiden.
- Repressor: Der Promotor reguliert die Genexpression über seine Zugänglichkeit für die RNA-Polymerase. Wenn der Promotor frei ist, kann die Polymerase an ihn binden und die Transkription kann ungehindert stattfinden. Jedoch können sogenannte Repressor-Proteine an den Promotor binden und so verhindern, dass er für die RNA-Polymerase zugänglich ist. Die Transkription wird dadurch unterbunden. Diese Regulation findet man häufig in Operon bei Bakterien.
- Aktivator: Manche Promotoren sind alleine „zu schwach“, um die RNA-Polymerase fest genug zu binden, so dass die Transkription nicht gestartet werden kann. Um die Bindung zwischen DNA und RNA-Polymerase am Promotor zu unterstützen, werden Aktivator-Proteine benötigt, welche ebenfalls an den Promotor binden. Nur wenn diese Proteine an die Promotor-Region gebunden haben, kann die Transkription des Gens ablaufen.
Das Modell, welches hier behandelt wird, wird genau diese beiden Typen beinhalten; einen Repressor und einen Aktivator für die Genexpressionsregulation.
3.5 – RNA-Polymerase
Um ein Gen abzulesen und eine RNA-Kopie der codierenden Region zu erstellen, benötigt es einen speziellen Enzym-Komplex: eine DNA-abhängige RNA-Polymerase. Wie der Name schon sagt, kann dieses Enzym anhand einer DNA-Vorlage eine RNA-Sequenz erstellen, welche komplementär zum DNA-Strang ist und Thymin durch Uracil ersetzt wurde (Abb. 3.3). Um diese komplexe Aufgabe zu erfüllen, ist dieser Enzymkomplex aus mehreren Untereinheiten aufgebaut, welche alle eine bestimmte Teilaufgabe erfüllen. Somit hat die RNA-Polymerase die Fähigkeit unterschiedliche Promotoren zu erkennen, an DNA zu binden und Nucleotide zu einem RNA Strang zu verknüpfen.
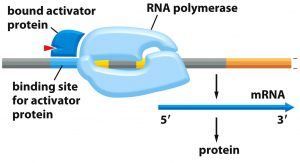
Abbildung 3.3 – Funktion der RNA-Polymerase. Die RNA-Polymerase bindet an die DNA, falls der Promotor aktiviert ist. Dies kann sein, wenn das Promotor-bindende Protein ihn aktiviert (wird in folgenden Kapiteln Protein A genannt) oder deaktiviert (wird in den folgenden Kapiteln Protein R genannt). Die Polymerase liest dann einen Strang und fügt komplementäre Nucleotide zu einer mRNA zusammen, welche dann im Prozess der Translation zu Protein führen. (Figure 8-8 von Essential Cell Biology, Garland Science, 2010)
3.6 – Transkription und Transkriptionswahrscheinlichkeit β:
Gene können im Prozess der Transkription in mRNA umgewandelt werden. Ihr Open-Reading-Frame wird dabei von einer RNA-Polymerase in mRNA kopiert. Somit kann die Information, die das Gen codiert, kurzfristig auf einem anderen Medium als DNA gespeichert werden. Bei Eukaryoten muss die mRNA den Zellkern verlassen, damit im Cytoplasma der Zelle die Protein-Synthese stattfinden kann. Da Prokaryoten keinen Zellkern besitzen, kann die Translation parallel zur Transkription stattfinden.
Dieser Prozess funktioniert nur, wenn der Promotor des Gens, welches transkribiert werden soll, aktiv ist. Allerdings ist es auch möglich, dass obwohl der Promotor aktiv ist, keine Polymerase bindet, da die Konzentration von freier RNA-Polymerase zu niedrig ist. Ein Mass dafür, wie wahrscheinlich es ist, dass ein Gen bei aktivem Promotor tatsächlich transkribiert wird, wird durch die Transkriptionswahrscheinlichkeit β beschrieben.
3.7 – Translation und Translationsrate m:
Im Cytoplasma der Zelle befinden sich Ribosomen, die „Protein-Baumeister“ der Zellen. Ribosomen sind makromolekulare Komplexe, die anhand der auf der mRNA gespeicherten Information das jeweilige Protein aus einzelnen Aminosäuren zusammenbauen können. Ribosomen können ausgehend von dieser mRNA-„Bauanleitung“ mehrere Moleküle des codierten Proteins herstellen. Wie viele Moleküle des Proteins im Durchschnitt aus einem mRNA-Strang hergestellt werden können, wird durch die Translationsrate [latex]m[/latex] beschrieben. Das Protein wird gegebenenfalls noch innerhalb der Zelle weitertransportiert, um seine Funktion am richtigen Ort ausführen zu können.
3.8 – Genexpression:
Wird aus dem Gen, also der Information, ein physischer Bestandteil einer Zelle, also eine funktionale Einheit, spricht man vom Prozess der Genexpression. Dies kann ein Protein sein, es gibt aber auch RNA-Moleküle wie tRNA und rRNA, welche ebenfalls funktionale Einheiten sind.
3.9 – Regulation der Genexpression:
Zellen sind sparsam, sie wollen nicht mehr Protein herstellen als notwendig. Gleichzeitig möchte die Zelle aber auch gut auf wechselnde Umwelteinflüsse reagieren können wie Nahrungsangebot, Dichte- oder Hitzestress. Die Zelle hat daher eine Reihe von Regulationsmechanismen, die in verschiedenen Stadien der Genexpression wirken können.
Ein interessanter Mechanismus auf Ebene der Transkription ist das negative Feedback. Negatives Feedback beschreibt im Allgemeinen einen Vorgang, bei dem das Produkt eines Prozesses hemmend auf den eigenen Entstehungsmechanismus rückwirkt. In diesem Buch wird folgendes Setting von einem negativen Feedback verwendet (Abb. 3.4):
- Ein Gen [latex]A[/latex] codiert das Protein A, welches auch exprimiert wird.
- Ein zweites Gen [latex]R[/latex] kodiert das Protein [latex]R[/latex], dessen Transkription von [latex]A[/latex] aktiviert wird, indem A an den Promotor [latex]R[/latex] binden kann. A ist somit ein Aktivator von [latex]R[/latex].
- R kann nun an den Promotor vom Gen [latex]A[/latex] binden, wo es die Transkription von [latex]A[/latex] verhindert. R ist somit ein Repressor von [latex]A[/latex], der sich somit auch negativ auf die Aktivierung der eigenen Expression auswirkt.
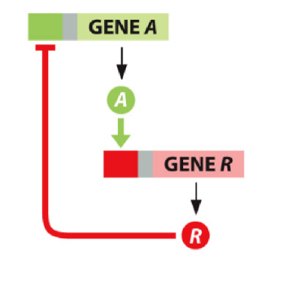
Abbildung 3.4 – Überblick des negativen Feedbackmechanismus. Protein A bindet an den Promotor von Gen R, welcher dadurch aktiviert wird. Dies führt zur Synthese des Proteins R. Dieses bindet dann an den Promotor von Gen A, welcher dadurch deaktiviert wird. Das Produkt von Gen A hemmt durch diesen Mechanismus die eigene Herstellung.
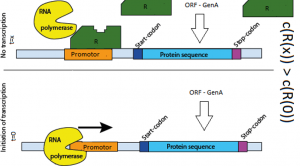
Abbildung 3.5 – Negatives Feedback für die Transkription. Unten: Wenn noch kein Repressor (R) vorhanden ist, kann die RNA-Polymerase am Promotor binden und die Transkription findet statt. Diese Transkription führt zur Herstellung eines Protein R, welches dann an den Promotor des Gens A bindet und die Transkription inhibiert. (Abbildung von promoters and operators, which combination is the best, DNAcoil.com, 09.06.2013)
Mit der Zeit werden sowohl A, als auch R abgebaut. Wie schnell das jeweilige Protein abgebaut wird, hängt von seiner Durchschnittlichen Lebensdauer [latex]\tau[/latex] ab.
Überlässt man ein System wie dieses sich selbst, kann sich mit der Zeit ein chemisches Gleichgewicht einstellen. Ein chemisches Gleichgewicht liegt hier also dann vor, wenn die Menge an Protein, die an den jeweiligen Promotor neu bindet, der Menge an Protein entspricht, die am Promotor abgebaut wird.